Hva er GLM i R?
Generaliserte lineære modeller er en undergruppe av lineære regresjonsmodeller og støtter ikke-normale distribusjoner effektivt. For å støtte dette anbefales det å bruke glm () -funksjonen. GLM fungerer bra med en variabel når variansen ikke er konstant og distribueres normalt. En koblingsfunksjon er definert for å transformere responsvariabelen slik at den passer til den aktuelle modellen. En LM-modell er gjort med både familien og formelen. GLM-modellen har tre viktige komponenter som kalles tilfeldig (sannsynlighet), systematisk (lineær prediktor), linkkomponent (for logit-funksjon). Fordelen med å bruke glm er at de har modellfleksibilitet, ikke behov for konstant varians, og denne modellen passer til maksimal sannsynlighetsestimering og dens forhold. I dette emnet skal vi lære om GLM i R.
GLM-funksjon
Syntaks: glm (formel, familie, data, vekter, delmengde, Start = null, modell = SANN, metode = ””…)
Her inkluderer familietyper (inkluderer modelltyper) binomial, Poisson, Gaussian, gamma, quasi. Hver distribusjon utfører en annen bruk og kan brukes i enten klassifisering og prediksjon. Og når modellen er gaussisk, bør responsen være et reelt heltall.
Og når modellen er binomial, bør responsen være klasser med binære verdier.
Og når modellen er Poisson, bør responsen være ikke-negativ med en numerisk verdi.
Og når modellen er gamma, bør responsen være en positiv numerisk verdi.
glm.fit () - For å passe til en modell
Lrfit () - angir passform for logistisk regresjon.
update () - hjelper til med å oppdatere en modell.
anova () - det er en valgfri test.
Hvordan lage GLM i R?
Her skal vi se hvordan lage en enkel generalisert lineær modell med binære data ved å bruke glm () -funksjon. Og ved å fortsette med Trees-datasettet.
eksempler
// Importere et biblioteklibrary(dplyr)
glimpse(trees)

For å se kategoriske verdier tildeles faktorer.
levels(factor(trees$Girth))
// Verifisere kontinuerlige variabler
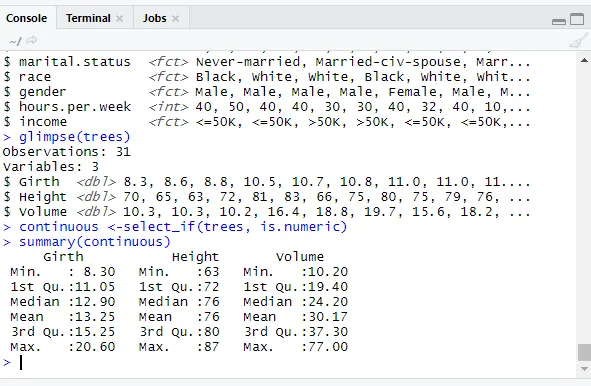
library(dplyr)
continuous <-select_if(trees, is.numeric)
summary(continuous)
// Inkludert tredatasett i R-søk Pathattach (trær)
x<-glm(Volume~Height+Girth)
x
Produksjon:
| Ring: glm (formel = Volum ~ Høyde + Omkrets)
koeffisienter: (Avskjæring) Høyde Omkrets -57.9877 0.3393 4.7082 Degrees of Freedom: 30 Total (dvs. Null); 28 Rest Null avvik: 8106 Restavvik: 421.9 AIC: 176.9 |
summary(x)
| Anrop:
glm (formel = Volum ~ Høyde + Omkrets) Avviksrester: Min 1Q Median 3Q Maks -6.4065 -2.6493 -0.2876 2.2003 8.4847 koeffisienter: Estimate Std. Feil t verdi Pr (> | t |) (Intercept) -57.9877 8.6382 -6.713 2.75e-07 *** Høyde 0.3393 0.1302 2.607 0.0145 * Omkrets 4.7082 0.2643 17.816 <2e-16 *** - Signif. koder: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0, 1 '' 1 (Dispersjonsparameter for gaussisk familie antatt å være 15.06862) Null avvik: 8106.08 på 30 frihetsgrader Restavvik: 421, 92 på 28 frihetsgrader AIC: 176, 91 Antall Fisher-iterasjoner: 2 |
Utgangen fra sammendragsfunksjonen gir ut samtaler, koeffisienter og rester. Ovennevnte svar viser at både høyde og omkrets er effektiv uten betydning, ettersom sannsynligheten for dem er mindre enn 0, 5. Og det er to varianter av avvik som heter null og gjenværende. Endelig er fiskerscoring en algoritme som løser maksimale sannsynlighetsproblemer. Med binomial er responsen en vektor eller matrise. cbind () brukes til å binde kolonnevektorene i en matrise. Og for å få detaljert informasjon om passformsammendraget blir brukt.
For å gjøre som hette-test utføres følgende kode.
step(x, test="LRT")
Start: AIC=176.91
Volume ~ Height + Girth
Df Deviance AIC scaled dev. Pr(>Chi)
421.9 176.91
- Height 1 524.3 181.65 6.735 0.009455 **
- Girth 1 5204.9 252.80 77.889 < 2.2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Call: glm(formula = Volume ~ Height + Girth)
Coefficients:
(Intercept) Height Girth
-57.9877 0.3393 4.7082
Degrees of Freedom: 30 Total (ie Null); 28 Residual
Null Deviance: 8106
Residual Deviance: 421.9 AIC: 176.9
Modell passform
a<-cbind(Height, Girth - Height)
> a
Sammendrag (trees)
Girth Height Volume
Min. : 8.30 Min. :63 Min. :10.20
1st Qu.:11.05 1st Qu.:72 1st Qu.:19.40
Median :12.90 Median :76 Median :24.20
Mean :13.25 Mean :76 Mean :30.17
3rd Qu.:15.25 3rd Qu.:80 3rd Qu.:37.30
Max. :20.60 Max. :87 Max. :77.00
For å få passende standardavvik
apply(trees, sd)
Girth Height Volume
3.138139 6.371813 16.437846
predict <- predict(logit, data_test, type = 'response')
Deretter henviser vi til telleresponsvariabelen for å modellere en god responspasning. For å beregne dette bruker vi USAccDeath datasettet.
La oss gå inn i følgende kodebiter i R-konsollen og se hvordan årstallet og årstorget utføres på dem.
data("USAccDeaths")
force(USAccDeaths)
// For å analysere året fra 1973-1978.
disc <- data.frame(count=as.numeric(USAccDeaths), year=seq(0, (length(USAccDeaths)-1), 1)))
yearSqr=disc$year^2
a1 <- glm(count~year+yearSqr, family="poisson", data=disc)
summary(a1)
| Anrop:
glm (formel = telle ~ år + årSqr, familie = “poisson”, data = plate) Avviksrester: Min 1Q Median 3Q Maks -22.4344 -6.4401 -0.0981 6.0508 21.4578 koeffisienter: Estimate Std. Feil z-verdi Pr (> | z |) (Intercept) 9.187e + 00 3.557e-03 2582.49 <2e-16 *** år -7.207e-03 2.354e-04 -30.62 <2e-16 *** årSkr 8.841e-05 3.221e-06 27.45 <2e-16 *** - Signif. koder: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0, 1 '' 1 (Dispersjonsparameter for Poisson-familien antatt å være 1) Null avvik: 7357, 4 på 71 frihetsgrader Restavvik: 6358, 0 på 69 frihetsgrader AIC: 7149.8 Antall Fisher-iterasjoner: 4 |
For å bekrefte at modellen passer best, kan følgende kommando brukes til å finne
restene til testen. Fra resultatet nedenfor er verdien 0.
1 - pchisq(deviance(a1), df.residual(a1))
Bruke QuasiPoisson-familien for større varians i de gitte dataene
a2 <- glm(count~year+yearSqr, family="quasipoisson", data=disc)
summary(a2)
| Anrop:
glm (formel = telle ~ år + årSqr, familie = “quasipoisson”, data = plate) Avviksrester: Min 1Q Median 3Q Maks -22.4344 -6.4401 -0.0981 6.0508 21.4578 koeffisienter: Estimate Std. Feil t verdi Pr (> | t |) (Intercept) 9.187e + 00 3.417e-02 268.822 <2e-16 *** år -7.207e-03 2.261e-03 -3.188 0.00216 ** årSkv. 8.841e-05 3.095e-05 2.857 0.00565 ** - (Dispersjonsparameter for quasipoisson-familie antatt å være 92.28857) Null avvik: 7357, 4 på 71 frihetsgrader Restavvik: 6358, 0 på 69 frihetsgrader AIC: NA Antall Fisher-iterasjoner: 4 |
Sammenligning av Poisson med binomial AIC-verdi er betydelig forskjellig. De kan analyseres med presisjons- og tilbakekallingsforhold. Neste trinn er å verifisere resterende varians er proporsjonal med gjennomsnittet. Da kan vi plotte ved å bruke ROCR-bibliotek for å forbedre modellen.
Konklusjon
Derfor har vi fokusert på spesiell modell kalt generalisert lineær modell som hjelper med å fokusere og estimere modellparametrene. Det er først og fremst potensialet for en kontinuerlig responsvariabel. Og vi har sett hvordan glm passer til en R innebygde pakker. De er de mest populære tilnærmingene for måling av telledata og et robust verktøy for klassifiseringsteknikker brukt av en dataforsker. R-språk hjelper selvfølgelig med å utføre kompliserte matematiske funksjoner
Anbefalte artikler
Dette er en guide til GLM i R. Her diskuterer vi GLM-funksjonen og Hvordan lage GLM i R med tredatasett eksempler og utdata. Du kan også se på følgende artikkel for å lære mer -
- R Programmeringsspråk
- Big Data Architecture
- Logistisk regresjon i R
- Big Data Analytics jobber
- Poisson Regresjon i R | Implementering av Poisson Regression